3. 分析流程与内容解读
免疫组库分析通常包含以下核心步骤,每一步都揭示了免疫应答的不同层面信息:
3.1 细胞质控
细胞质控是确保单细胞免疫组库分析准确可靠的基石。此步骤旨在通过一系列严格的质量评估指标,过滤掉低质量的细胞和测序数据,为后续的生物信息学分析提供高质量的输入。为了保证分析结果的可靠性,我们关注以下核心质控指标:
- 文库质量:合格的 TCR/BCR 文库在 Agilent 4200 TapeStation 检测时,应在 250-1500bp 范围内呈现特异性尖峰,且文库浓度不低于 1ng/μL。
- 测序深度:建议每个细胞的平均测序 Reads 数(Mean Read Pairs per Cell)不低于 5,000,以确保能够捕获到足够的信息进行准确的 Contig 拼接。
- 数据质量:
- Estimated Number of Cells:评估捕获到的细胞数量。
- Valid Barcodes:带有有效细胞 Barcode 的 Reads 比例,通常应 > 80%。
- Q30 Bases in Barcode/UMI:Barcode 和 UMI 序列中,测序质量值大于等于 Q30 的碱基比例,是衡量测序准确性的关键指标,通常要求 > 90%。
- Reads Mapped to Any V(D)J Gene:比对到 V(D)J 参考基因的 Reads 比例,反映了 V(D)J 富集的特异性和效率。
- Number of Cells With Productive V-J Spanning Pair:鉴定出具有完整且有功能的 V-J 配对链的细胞数量,这是后续克隆型分析的基础。
3.2 免疫组库拼接与克隆型定义
此步骤的目标是将原始测序数据转化为具有生物学意义的免疫细胞克隆,核心流程包括TCR/BCR结构识别、序列组装注释与最终的克隆型定义。
3.2.1 TCR/BCR 结构简介
TCR 和 BCR 的结构决定了其识别抗原的特异性。其可变区由 V、D、J 基因片段重排形成,其中 CDR3(互补决定区3) 是变异最大的区域,直接决定了抗原结合的特异性。
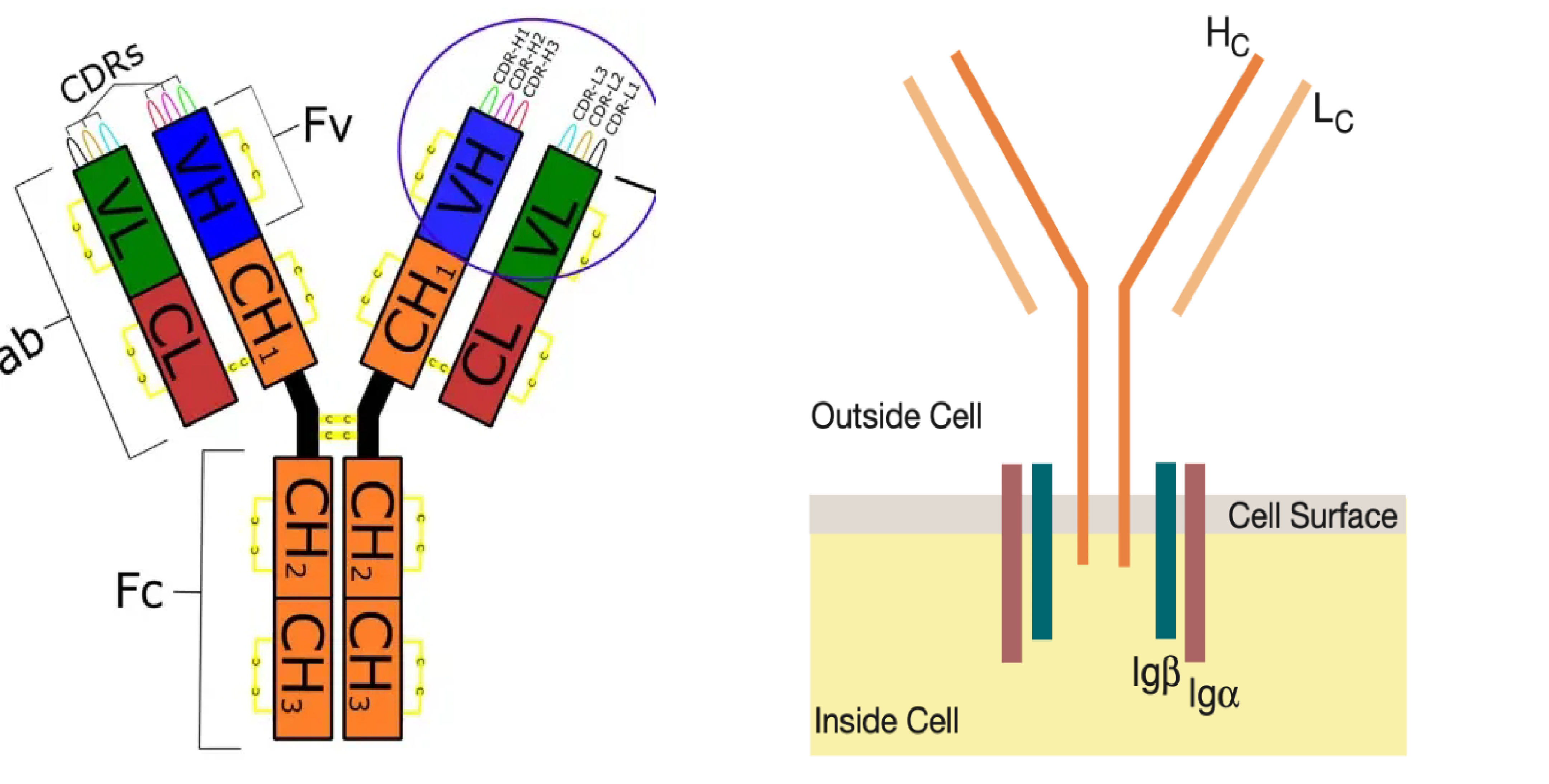 BCR 由两条重链(H)和两条轻链(L)组成,其抗原结合能力由 V 区决定。
BCR 由两条重链(H)和两条轻链(L)组成,其抗原结合能力由 V 区决定。
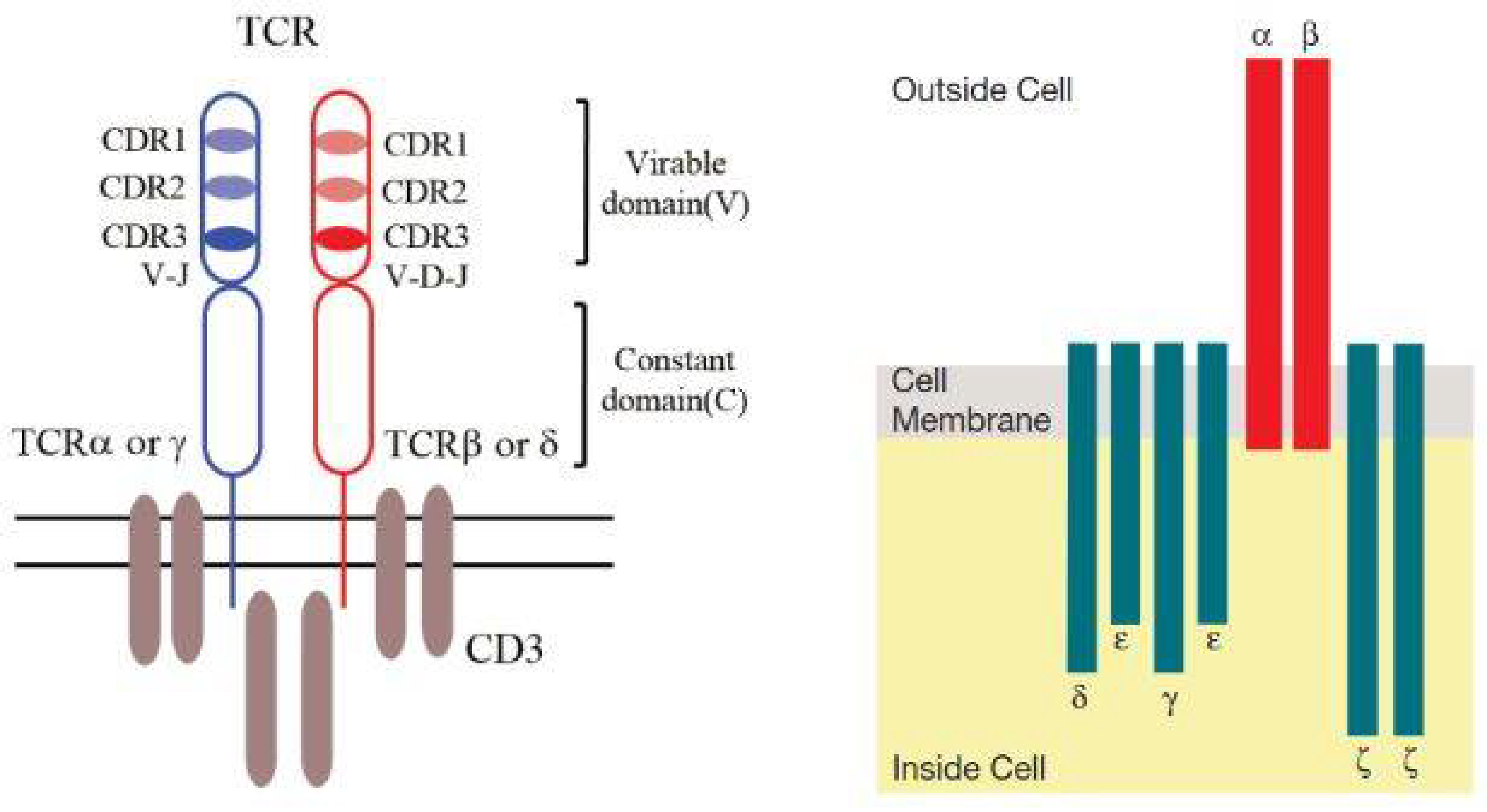 TCR 主要为 αβ 链或 γδ 链组成的异二聚体,通过可变区识别并结合由 MHC 分子递呈的抗原肽。
TCR 主要为 αβ 链或 γδ 链组成的异二聚体,通过可变区识别并结合由 MHC 分子递呈的抗原肽。
3.2.2 序列组装与 VDJ 注释
SeekSoul® Tools 采用以下流程进行序列组装与注释:
- 序列提取与降采样:利用 TRUST4 工具从原始数据中提取可能包含 TCR/BCR 序列的 Reads。为平衡计算效率,对于 Reads 数超过 80,000 的细胞,会进行降采样处理。
- UMI 矫正与序列组装:在组装前,使用
umitools对 UMI 进行矫正,以消除测序和 PCR 扩增引入的错误。随后,基于矫正后的 UMI 和 Reads,TRUST4 对每个细胞的 V(D)J 序列进行组装,生成 Contigs。 - VDJ 基因注释:利用 TRUST4 的
annotator模块对组装好的 Contigs 进行注释,确定 V/D/J/C 基因的使用情况以及 CDR3 的精确位置和序列。- Primary Chain 选择:在一个细胞内,如果某条链(如 TRB)有多条注释结果,软件会选择 UMI 数量最高 的那条 Contig 作为该链的
primary_chain。
- Primary Chain 选择:在一个细胞内,如果某条链(如 TRB)有多条注释结果,软件会选择 UMI 数量最高 的那条 Contig 作为该链的
3.2.3 Clonotype 定义
Clonotype(克隆型)是一组具有相同或高度相似免疫受体的细胞。SeekSoul® Tools 通过以下精细的步骤来定义克隆型:
- 高质量 Contig 筛选:首先,对细胞进行筛选,要求其支持 Contig 的 UMI 总数不少于 3。
- 基于 CDR3 序列相似性聚类:使用 dandelion 工具,基于 CDR3 氨基酸序列 的相似性来定义克隆。相似性阈值根据受体类型有所不同:
- BCR 设置为 85%
- TCR 设置为 100%
- 单链克隆型的优化处理:为优化单链克隆型(如仅含TRB链),软件会自动将其与具有相同VJ基因及CDR3序列的双链克隆型进行匹配与合并,以确保克隆型定义的准确性。
TIP
Contig 拼接的质量和 Clonotype 定义的准确性直接决定了后续所有分析的可靠性。在分析报告中,应重点关注高质量(high_confidence, full_length, productive)Contig 的数量和最终鉴定出的克隆型数量。
3.3 细胞映射
此步骤的目标是将VDJ库(克隆型身份)与GEX库(转录组功能)在单细胞水平上进行精确匹配,从而将细胞的克隆型特征与其功能状态关联起来。
3.3.1 映射原理
由于 VDJ 库和 5' GEX 库共享同一套细胞 Barcode 系统,我们可以精确地将每个细胞的 TCR/BCR 序列信息与其转录组表达谱关联起来。这使得我们能够在特定的细胞亚群(如耗竭性 T 细胞、记忆 B 细胞)中研究其免疫受体的特征。
3.3.2 映射结果解读
通过 UMAP 等降维可视化方法,可以直观地展示 VDJ 信息在不同细胞亚群中的分布情况。
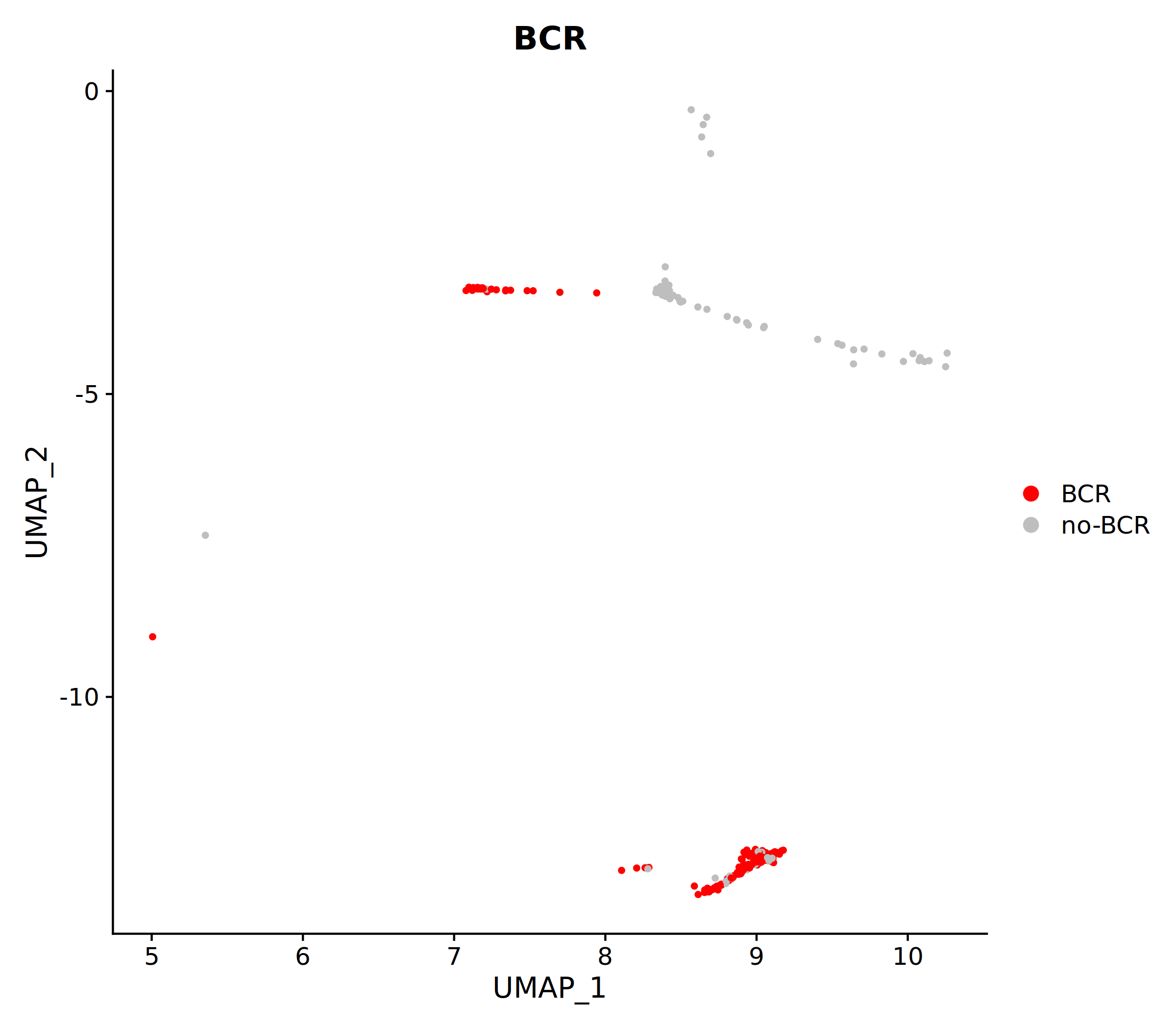 上图展示了 BCR 检出细胞(红色)在 UMAP 聚类图中的分布。通过观察红色细胞主要富集在哪些细胞群中,可以分析不同 B 细胞亚群的 BCR 检出率及其生物学意义。
上图展示了 BCR 检出细胞(红色)在 UMAP 聚类图中的分布。通过观察红色细胞主要富集在哪些细胞群中,可以分析不同 B 细胞亚群的 BCR 检出率及其生物学意义。
- "TCR/BCR" 细胞:在 VDJ 库中成功检测到免疫受体序列的细胞。
- "no-TCR/BCR" 细胞:未检测到免疫受体序列的细胞。可能的原因包括:
- 该细胞本身不表达 TCR/BCR(如巨噬细胞、成纤维细胞)。
- 免疫受体基因表达水平极低,低于检测阈值。
- 细胞裂解不充分或 V(D)J 富集效率不佳。
NOTE
细胞映射的检出率是衡量实验质量的重要指标之一。较低的检出率可能意味着需要优化实验条件或提高测序深度。通过分析特定细胞类型的检出率,可以为后续的功能研究提供重要线索。
3.4 V/J 基因特征分析
V/J 基因的使用频率和配对模式是免疫组库的关键特征,反映了免疫系统对抗原的响应偏好和克隆选择。通过分析这些特征,可以深入了解免疫应答的广度与特异性。
3.4.1 V/J 基因使用频率 (Gene Usage)
该分析统计 V 基因和 J 基因在免疫组库中的使用频率。在免疫应答过程中,某些 V/J 基因可能因为能有效识别特定抗原而被优先选择和扩增,导致其在组库中的占比显著高于其他基因。
- 分析方法:分别对整体和每组中每个细胞类型的 V 基因和 J 基因丰度分布情况进行统计。
- 结果解读:通常使用柱状图展示各个 V/J 基因的相对丰度。图中 X 轴代表不同的 V 或 J 基因,Y 轴代表其在总克隆型中的百分比(frequency)。优势表达的基因(即较高的柱子)可能与样本所处的特定免疫状态相关。
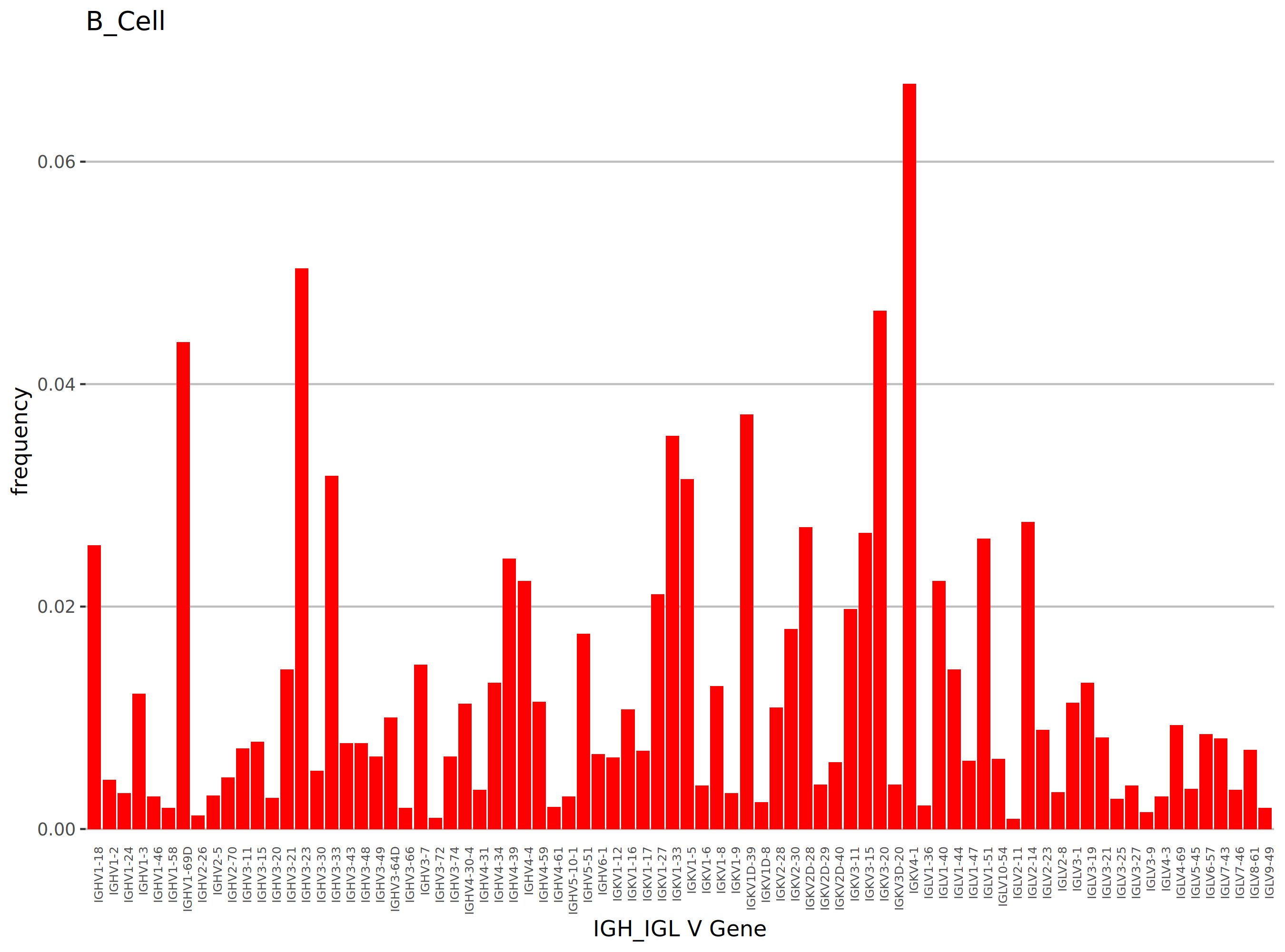 以BCR为例,TCR分析同理。上图展示了 B 细胞中不同 V 基因的使用频率分布,可直观识别丰度最高的优势基因。横坐标为 IGH_IGL V 基因名称,纵坐标为频率。
以BCR为例,TCR分析同理。上图展示了 B 细胞中不同 V 基因的使用频率分布,可直观识别丰度最高的优势基因。横坐标为 IGH_IGL V 基因名称,纵坐标为频率。
TIP
解读要点:
- 通过比较不同细胞类型或不同样本间的 V/J 基因使用模式,可以识别特定的免疫应答特征。
- 显著富集的 V/J 基因可能提示存在针对特定抗原的克隆扩增。
- 结合后续的差异分析热图,可以更全面地评估细胞类型间的 V/J 基因使用差异。
3.4.2 V/J 基因差异分析
为了更系统地比较不同细胞类型间的 V/J 基因使用差异,可以对整体和每组中细胞类型间的 V/J 基因频率进行差异分析。
- 结果解读:热图中每一行代表一个 V 或 J 基因,每一列代表一个细胞类型。颜色编码表示基因使用频率的标准化得分(Z-Score),橙色/红色表示该基因在该细胞类型中使用频率较高(上调),蓝色表示使用频率较低(下调)。通过层次聚类,可以识别具有相似 V/J 基因使用模式的细胞类型群组。
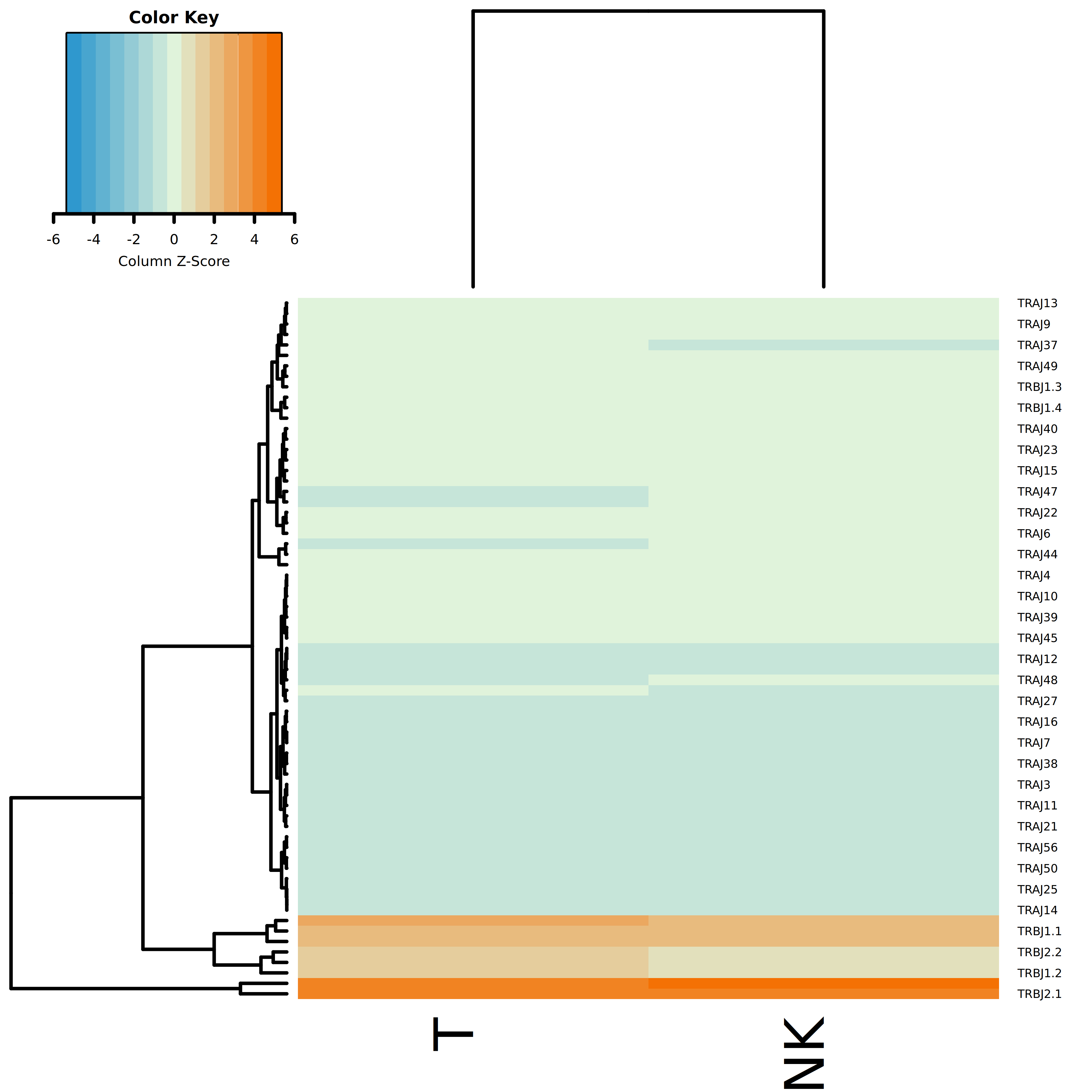 上图展示了不同细胞类型间 J 基因使用频率的差异分析热图。通过颜色深浅和聚类树可以识别细胞类型特异性的基因使用模式。
上图展示了不同细胞类型间 J 基因使用频率的差异分析热图。通过颜色深浅和聚类树可以识别细胞类型特异性的基因使用模式。
3.4.3 V-J 基因配对分析
除了单个基因的使用频率,V 和 J 基因的配对组合也包含重要的生物学信息。某些特定的 V-J 基因组合可能更倾向于被选择,形成优势配对,这同样暗示了其在抗原识别中的重要作用。
- 分析方法:在不同免疫组库中统计 V-J 基因对的配对频率,可以反映 CDR3 或免疫组库的变化特征。通过比较不同免疫时期高丰度的 V-J 基因对,可以识别特异性表达的免疫基因组合。
- 结果解读:V-J 配对信息常通过和弦图(Chord Diagram)进行可视化。和弦图能够直观展示不同 V 基因(外环的一侧)和 J 基因(外环的另一侧)之间的连接关系,连接条带的宽度与该 V-J 配对的丰度成正比。条带越宽,表示该配对组合在样本中的出现频率越高。
 上图通过 V-J 基因配对和弦图,清晰地展示了 T 细胞中优势 V-J 配对组合及其相对丰度。外环显示不同的 V 和 J 基因,连线宽度代表配对频率。
上图通过 V-J 基因配对和弦图,清晰地展示了 T 细胞中优势 V-J 配对组合及其相对丰度。外环显示不同的 V 和 J 基因,连线宽度代表配对频率。
3.5 CDR3 特征分析
CDR3 区是 TCR/BCR 可变区的核心,直接决定了抗原结合的特异性。该区域由 V、D、J 基因片段共同编码,并通过随机的核苷酸插入和删除形成极高的多样性。因此,对 CDR3 的特征分析是免疫组库研究的重中之重。
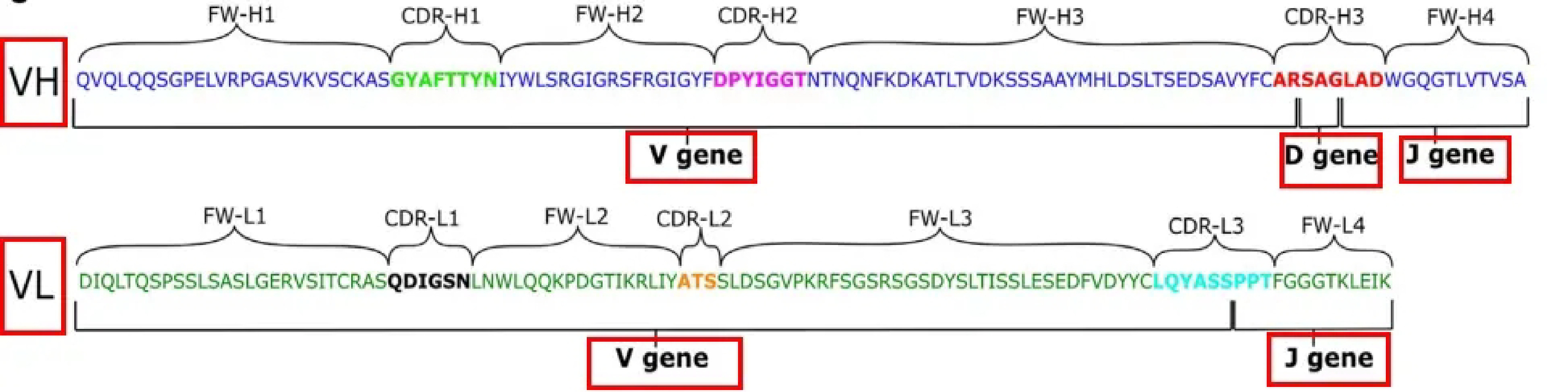 BCR 和 TCR 的可变区包含三个互补决定区(CDR1/CDR2/CDR3),其中 CDR3 的变异最大,直接决定了抗原结合的特异性。
BCR 和 TCR 的可变区包含三个互补决定区(CDR1/CDR2/CDR3),其中 CDR3 的变异最大,直接决定了抗原结合的特异性。
3.5.1 CDR3 序列长度与 V 基因组合分析
分析 CDR3 与 V 基因的组合特征,可以进一步揭示克隆选择的规律。通过"谱系图"(Spectratype Plot)可以同时展示 CDR3 长度分布与 V 基因使用频率的关系。
- 分析方法:对整体和每组中不同 CDR3 序列长度下 V 基因的分布情况进行统计。
- 结果解读:在谱系图中,X 轴为 CDR3 的碱基序列长度(bp),Y 轴为频率(frequency)。每个柱子代表特定长度的 CDR3,柱内的不同颜色分段代表构成该长度 CDR3 的不同 V 基因来源及其比例。图中对丰度前12的 V 基因用不同颜色填充,其余为灰色(Other)。这有助于识别是否存在特定长度的 CDR3 与特定 V 基因的优势组合。
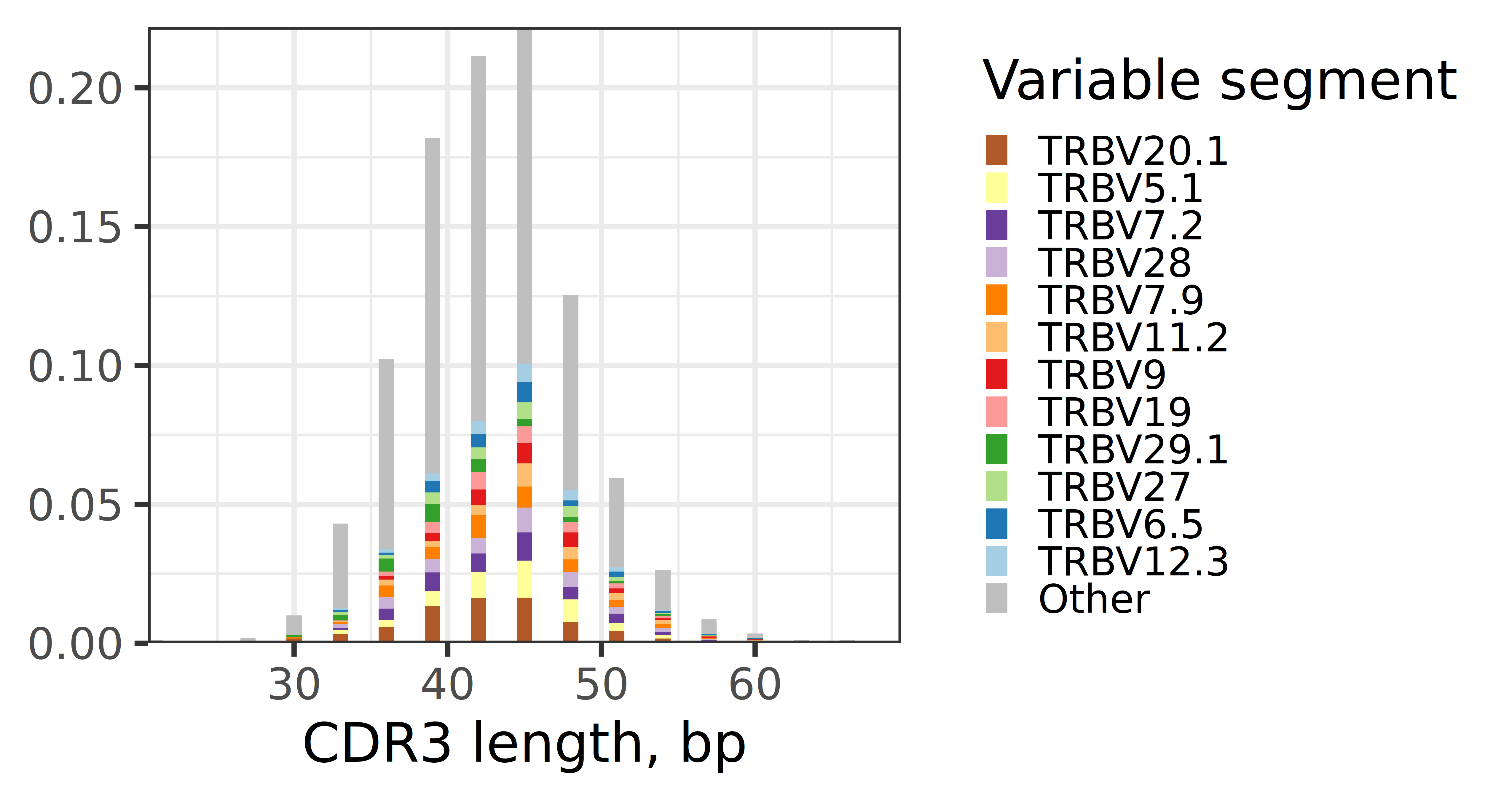 上图展示了 T 细胞中不同 CDR3 长度下,各类 V 基因的分布情况。横坐标为 CDR3 碱基序列长度,纵坐标为频率,不同颜色代表不同的 V 基因(TRBV)。通过此图可以分析特定 V 基因是否倾向于形成特定长度的 CDR3。
上图展示了 T 细胞中不同 CDR3 长度下,各类 V 基因的分布情况。横坐标为 CDR3 碱基序列长度,纵坐标为频率,不同颜色代表不同的 V 基因(TRBV)。通过此图可以分析特定 V 基因是否倾向于形成特定长度的 CDR3。
NOTE
生物学意义:
- CDR3 长度与 V 基因的组合模式反映了免疫受体的结构特征和多样性。
- 某些 V 基因可能倾向于产生特定长度范围的 CDR3,这与其编码区的结构特点有关。
- 异常的 CDR3 长度分布或 V 基因偏好性可能提示存在克隆扩增或免疫选择。
3.6 CDR3 长度分布统计
CDR3 是 V(D)J 基因编码的核心区域,通常包含 V 基因的一部分、全部 D 基因以及部分 J 基因,因此是 BCR/TCR 上最具代表性、最具辨识度的区域。在绝大多数免疫研究中,会把 CDR3 序列作为定义和识别某一个特定 BCR 或 TCR 的唯一依据,即具有相同 CDR3 序列的细胞为一个克隆型。CDR3 序列的长度并非完全随机,其分布特征可以反映免疫组库的整体状态和克隆扩增程度。
3.6.1 CDR3 长度分布的生物学意义
在正常的免疫系统中,CDR3 长度通常呈现一种准高斯(bell-shaped)分布,反映了免疫组库的多样性和随机性。
- 正常分布:多样化的免疫组库通常表现为覆盖多种长度、形状对称的钟形曲线,提示免疫系统具有广泛的抗原识别能力。
- 分布偏移或寡克隆优势:当机体针对特定抗原(如病毒感染或肿瘤)产生强烈的免疫应答时,少数高效识别抗原的 T/B 细胞会大量扩增。这会导致其对应的 CDR3 长度在分布图上形成一个或几个突出的尖峰,使整体分布偏离标准形态。因此,CDR3 长度分布是评估克隆扩增程度的直观指标。
3.6.2 CDR3 长度分布分析方法
分别对整体和每组中每个细胞类型的 CDR3 序列长度分布情况进行统计。
- 柱状图展示:横坐标为 CDR3 序列长度,纵坐标为细胞数(counts),可以直观显示每个长度的绝对数量。
- 折线图展示:横坐标为 CDR3 序列长度(CDR3 Length),纵坐标为占比(Percent),可以清晰比较不同细胞类型或样本间的 CDR3 长度分布模式。
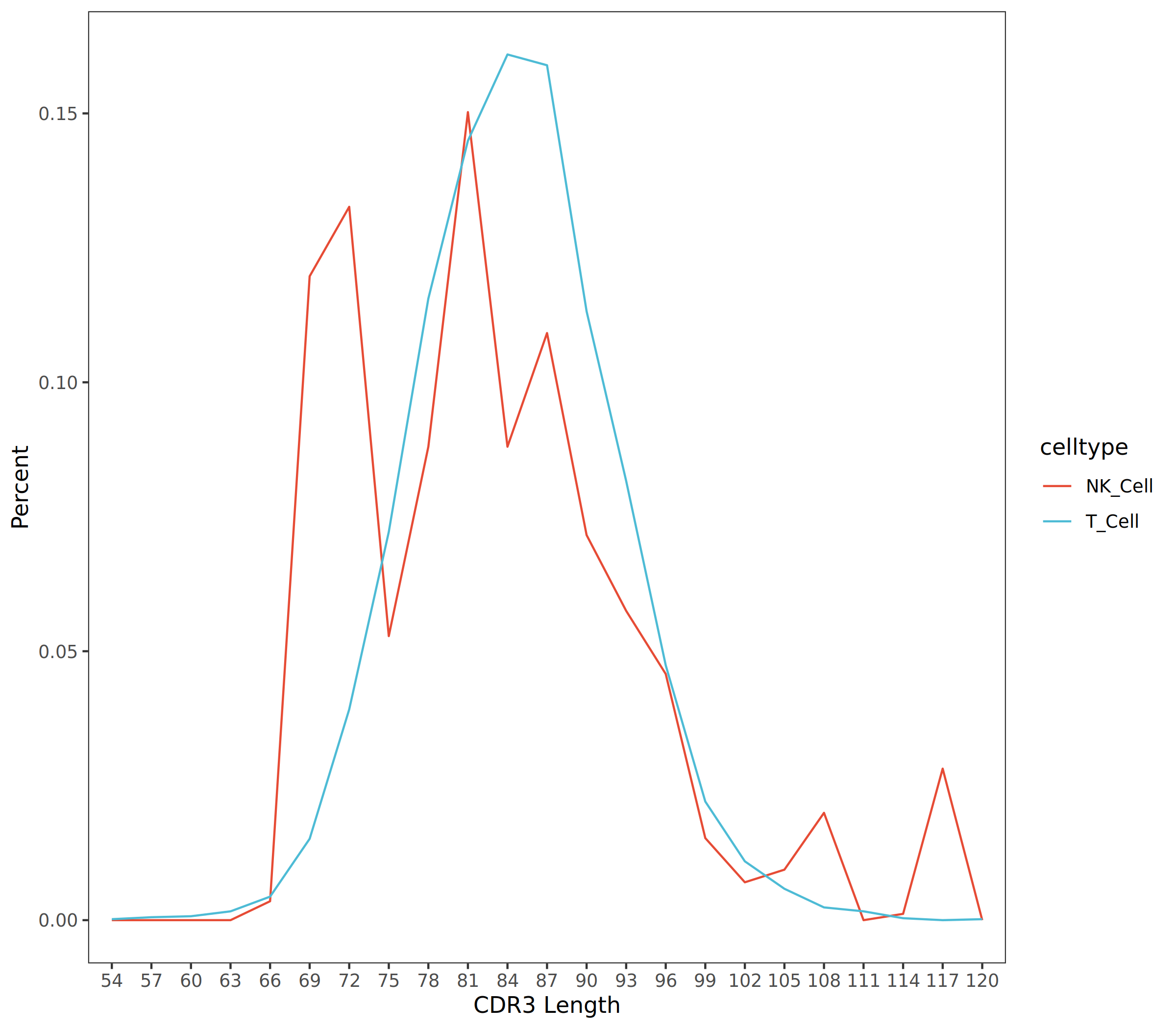 上图展示了不同细胞群(T_Cell 和 NK_Cell)中 CDR3 的长度分布情况。横坐标为 CDR3 长度,纵坐标为百分比。通过比较不同细胞群的分布曲线,可以评估各细胞群的克隆扩增状态和免疫组库多样性。T 细胞和 NK 细胞呈现出不同的 CDR3 长度分布特征,T 细胞的峰值主要集中在81-87bp左右,而 NK 细胞则在72和81bp附近有两个峰值。
上图展示了不同细胞群(T_Cell 和 NK_Cell)中 CDR3 的长度分布情况。横坐标为 CDR3 长度,纵坐标为百分比。通过比较不同细胞群的分布曲线,可以评估各细胞群的克隆扩增状态和免疫组库多样性。T 细胞和 NK 细胞呈现出不同的 CDR3 长度分布特征,T 细胞的峰值主要集中在81-87bp左右,而 NK 细胞则在72和81bp附近有两个峰值。
TIP
结果解读要点:
- 对称的钟形曲线提示正常的免疫组库多样性。
- 显著的尖峰或峰值偏移可能提示存在克隆扩增或免疫选择。
- 不同细胞类型可能具有不同的 CDR3 长度分布特征,这与其功能状态和发育阶段有关。
- 测定特定 CDR3 序列出现的频率可以反映免疫细胞扩增的程度,为后续的克隆型分析提供基础。
